Mainzelliste
Produktdetails
Produktdetails
Beschreibung
Die Mainzelliste ist ein webbasierter Pseudonymisierungsdienst und wurde als Nachfolger des PID-Generators der TMF entwickelt. Sie erlaubt die Erzeugung von Personenidentifikatoren (PID) aus identifizierenden Attributen (IDAT), dank Record-Linkage-Funktionalität auch bei wechselnder Qualität identifizierender Daten. Ihre Funktionen werden über eine REST-Schnittstelle bereitgestellt, die besonders flexible Integration durch andere Software ermöglicht.
Produktbroschüre "MAGIC": IT-Werkzeuge für die medizinische Verbundforschung
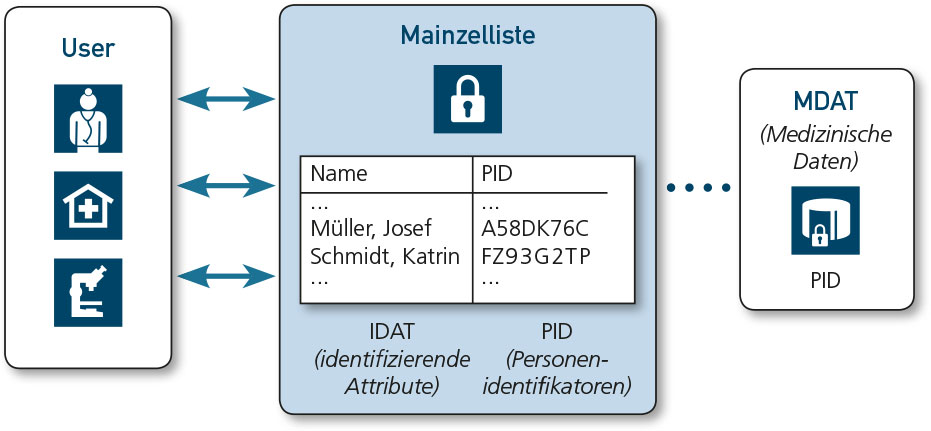
Erstellung nichtsprechender Pseudonyme (PID-Generierung)
Die Mainzelliste erzeugt für jeden eingetragenen Patienten einen oder mehrere nichtsprechende sogenannte Personenidentifikatoren (PID), die kompatibel zu denen des ursprünglichen PID-Generators sind. Diese deterministisch erzeugten achtstelligen Zeichenketten eignen sich sowohl für den Webeinsatz als auch manuelle Übertragung, da sie bis zu zwei Tippfehler erkennen können.
Record Linkage
Für jeden Patienten soll auch bei mehrmaliger Eingabe nur genau ein Pseudonym erzeugt werden. Dazu wird bei Anlage eines Patienten geprüft, ob dieser schon in der Datenbank vorhanden ist. Dank eines modularen Record-Linkage-Systems, das mit Hilfe einer Konfigurationsdatei flexibel an die Anforderungen des konkreten Anwendungsfalls angepasst werden kann, gelingt dies auch bei abweichender Schreibweise oder Vertippen. Neu im Vergleich zum PID-Generator ist insbesondere die Möglichkeit, eigene phonetische Codes sowie Zeichenkettenvergleiche zu nutzen, wodurch auch Namen aus anderen Sprachräumen fehlertolerant verglichen werden können. Derzeit wird ein gewichtsbasiertes Record Linkage unterstützt, die modulare Konzeption erlaubt aber auch die Nachrüstung eigener Algorithmen. Die Möglichkeit, unsicherere Zuordnungen manuell nachzubearbeiten, unterstützt darüber hinaus das automatische Matchverfahren.
Im Rahmen des Deutschen Konsortiums für Translationale Krebsforschung (kurz DKTK) wird außerdem basierend auf der Mainzelliste ein förderiertes Record Linkage betrieben. Das Record Linkage wird hierbei auf Basis von Bloomfiltern durchgeführt (Privacy-Preserving Record Linkage). Hierbei betreibt der Standort Frankfurt die Software zum Erzeugen der Bloomfilter und der Standort Mainz die Patientenliste.
REST-basierte Webschnittstelle
Eine leichtgewichtige REST-basierte Schnittstelle erlaubt eine einfache Anbindung unterschiedlichster Systeme, also z.B. Registern, Biobanken, EDC- und Studienmanagementsystemen. Das macht überhaupt erst die Verwendung aus Webbrowsern über AJAX- und JSONP-Aufrufe möglich. Implementierungen existieren für eine Vielzahl von Systemen, bspw. die Studienmanagementsysteme SecuTrial der Firma iAS, OpenClinica und Redcap.
Nutzer- und Administrationsschnittstelle
Die Komplexität des Record Linkage bleibt dem Nutzer verborgen, da er identifizierende Patientendaten über eine leicht verständliche, schlanke Bedienoberfläche in seinem Webbrowser eingibt.
Etwaige Fehler des Record Linkage können in einer administrativen Bedienoberfläche korrigiert werden (ab Version 1.11). Entsprechende Meldefunktionen vereinfachen den Prozess für Nutzer und Administratoren.
Abwärtskompatibilität
Für Verbünde, die noch den PID-Generator einsetzen, steht in den allermeisten Fällen ein Migrationspfad auf die Mainzelliste zur Verfügung. Dafür wurde die Phonetik nach dem Phonet-Algorithmus von Jörg Michael im Rahmen einer Evaluation in Java reimplementiert.
Die Mainzelliste selbst folgt einem kontrollierten Releasezyklus und ist seit ihrer ursprünglichen Version 1.0 (Mai 2013) abwärtskompatibel.
Zusammenführung verschiedener Datenklassen in Webbrowsern
In manchen behandlungsnahen Anwendungen ist es zulässig und wünschenswert, dass Nutzer anstelle von Pseudonymen Klarnamen sehen. Die REST-Schnittstelle der Mainzelliste ermöglicht prinzipiell deren Abruf, aber einige moderne und fast alle betagten Webbrowser halten für eine stabile Implementierung diverse Hürden bereit, u.a. die Same-Origin-Policy. Die Mainzelliste hilft diese Hindernisse zu umschiffen, indem eine Bibliothek für den Zugriff auf die Schnittstelle bereitgestellt wird.
Blocking und Locality Sensitive Hashing (ab v1.9)
Im Rahmen der „FASTML“-Förderung des TMF e.V. wurde das Bloomfilter-Matching der Mainzelliste dramatisch beschleunigt. Zur Anwendung kommt hierfür Locality-Sensitive Hashing. Eine Publikation belegt Geschwindigkeit und Matchgüte (Franke, 2018).
Audittrail (ab v1.9)
Zur Erfüllung von Anforderungen im Bereich klinischer Studien wurde die Mainzelliste um eine Audittrail-Funktionalität erweitert.
Secure Multi-Party Computation (Prototyp auf Github, Integration geplannt)
Der Mainzelliste Secure EpiLinker (MainSEL) stellt die nächste Stufe des Privacy-Preserving Record Linkage dar: Er ermöglicht Record Linkage und Pseudonymisierung zwischen zwei Mainzellisten, ohne dabei Bestandteile der IDAT preiszugeben (experimentell; siehe u.a. Publikation).
Weitere an der Entwicklung beteiligte Einrichtungen
- Andreas Borg, Universitätsmedizin Mainz
- Benjamin Gathmann, Universitätsklinikum Freiburg
- Christian Koch, DKFZ-Heidelberg
- Cornelius Knopp, Universitätsmedizin Göttingen, Handling von eGK-Nummern
- Daniel Volk, Universitätsmedizin Mainz
- Dirk Langner, Universitätsmedizin Greifswald
- Florens Rohde, Universität Leipzig, Blocking- and Locality Sensitive Hashing
- Florian Stampe, DKFZ-Heidelberg
- Galina Tremper, DKFZ-Heidelberg
- Jens Schwanke, Universitätsmedizin Göttingen
- Jürgen Riegel, Universitätsmedizin Mainz
- Martin Lablans, DKFZ-Heidelberg
- Matthias Lemmer, Universität Marburg, Audittrail
- Maximilian Ataian, Universitätsmedizin Mainz
- Moanes Ben Amor, DKFZ-Heidelberg
- Phillip Schoppmann, TU Darmstadt, MainSEL
- Project FP7-305653-chILD-EU
- Sebastian Stammler, TU Darmstadt, MainSEL
- Stephan Rusch, Universitätsklinikum Freiburg
- Tobias Kussel, TU Darmstadt, MainSEL
- Torben Brenner, DKFZ-Heidelberg
- Ziad Sehili, Universität Leipzig
Das Produkt im Einsatz
- Institut für Medizinische Informatik, Universität Münster
Zusätzliche Anwendungsbereiche
- Deutsches Krebsforschungszentrum (DKFZ), Heidelberg
Unterstützung
Support für die Mainzelliste gibt es auf Bitbucket durch die Entwicklungscommunity. Außerdem kann Kontakt über die E-Mail Adresse info@mainzelliste.de aufgenommen werden
Referenzen
Mainzelliste-Tutorial beim TMF-Tutorialtag im Rahmen des Programmpunktes
"MAGIC: IT-Werkzeuge für die medizinische Verbundforschung" am 19. März 2019 in Bonn.
Download Vortrag [PDF | 2 MB]
Vorstellung Mainzelliste beim TMF-Workshop "MAGIC" am 18. September 2018 in Berlin.
Download Vortrag [PDF | 1 MB]
Lablans M, Borg A, Ückert F: A RESTful interface to pseudonymization services in modern web applications. BMC Medical Informatics and Decision Making201515:2.
Faldum A., Pommerening K., An optimal code for patient identifiers. Computer methods and programs in biomedicine, 2005. 79: p. 81-8.
Fielding R.T., Architectural Styles and the Design of Network-based Software Architectures, in Building, R.N. Taylor, Editor. 2000, Citeseer. p. 162.
Michael J., Doppelgänger gesucht – Ein Programm für kontextsensitive phonetische Textumwandlung. c’t Magazin für Computertechnik, 1999. 25.
Warnecke T., Borg A., Ückert F., Lablans M., Fehlertolerantes Record Linkage von Patientendaten durch den Phonet-Algorithmus
Lablans, M., et al. Eine generische Softwarebibliothek zur Umsetzung des TMF-Datenschutzkonzepts A im Webeinsatz. in 55. Jahrestagung der Deutschen Gesellschaft für Medizinische Informatik, Biometrie und Epidemiologie (gmds). 2010.
Borg A., Lablans M., Ückert F., Mainzelliste.Client - Eine Bibliothek für den Zugriff auf Patientenlisten. in 60. Jahrestagung der Deutschen Gesellschaft für Medizinische Informatik, Biometrie und Epidemiologie (gmds). 2015
Franke M., Sehili Z., Rahm E., Parallel Privacy-Preserving Record Linkage using LSH-based Blocking. In: Proceedings of the 3rd International Conference on Internet of Things, 2018.
Rohde, F., Franke, M., Sehili, Z., Lablans, M., Rahm, E., 2021. Optimization of the Mainzelliste software for fast privacy-preserving record linkage. Journal of Translational Medicine 19, 33. https://doi.org/10.1186/s12967-020-02678-1

Kommentare
Um einen Kommentar verfassen zu können, müssen Sie sich zunächst anmelden!